Remerciement à David Koes
David Koes est la personne qui a développé SMINA, logiciel de docking grâce auquel le projet emolgine a pu voir le jour. Je tiens à le remercie chaleureusement d’avoir mis son code à disposition.
Projet ICOA beta-D-xylosidase
Recherche dans Ambinter de molécules ayant de l’affinité pour le site de la beta-D-xylopyranose dans b-D-xylosidase. (Version 1.0)
Fait le 13 /02/2025 par Pascal KREZEL
Introduction
Etape_1: Génération de molécules après repositionnement de fragments
Etape_2: Recherche de molécules similaires dans Ambinter
Etape_3: Analyse de molécules de la Chembl
Introduction
L’équipe du Dr Pierre Lafitte (ICOA, Orléans, France) travaille sur la beta-D-xylosidase. Les références de cette protéine sont les suivantes:
-UniProt: B5YB78,
-Chembl: ‘CHEMBL4658’, ‘CHEMBL4728’
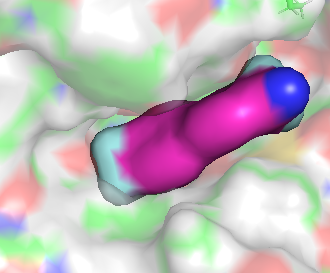
Les 2 poses cristallographiques présentent dans la PDB (Protein Data bank) sont issues du travail de son équipe. Les images suivantes sont issues du pdb 6YYI.
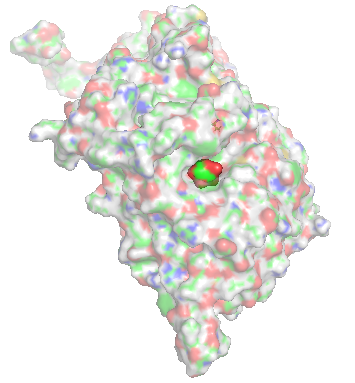 | 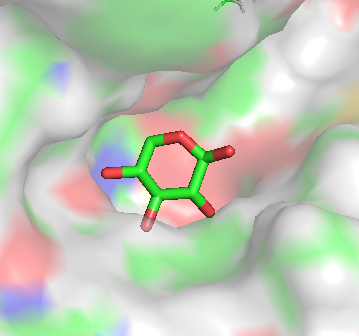 |
La minimisation de ce ligand donne le résultat suivant:
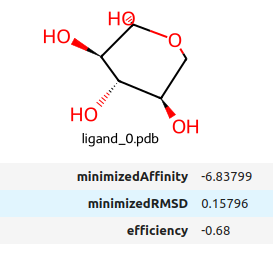
-0.68 comme valeur d’efficiency est une valeur très faible, indiquant que la molécule se positionne très bien.
Etape_1:Génération de molécules après repositionnement de fragments
La génération de molécules à partir de 10 000 fragments en utilisant les 1 000 meilleurs comme point de départ pour le growing a permis d’obtenir 159 528 molécules différentes avec une valeur de minimizedAffinity (score de SMINA) inférieure à -6 et une efficiency (minimizedAffinity divisé par le nombre d’atomes) inférieure à -0.25. Ce qui donne le graphique suivant:
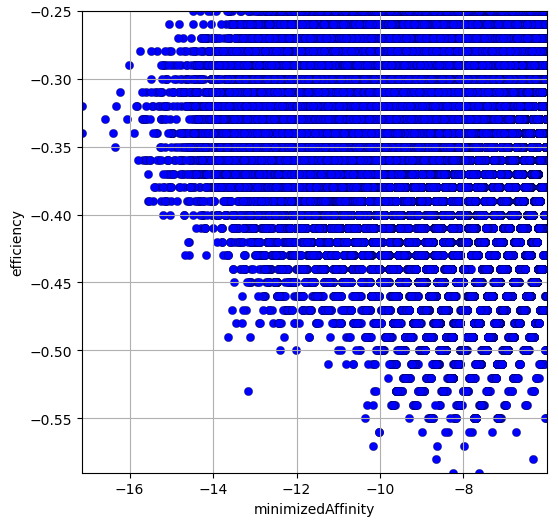
On constate qu’il est possible d’obtenir des solutions avec des valeurs très faibles (< -16) comme les suivantes:
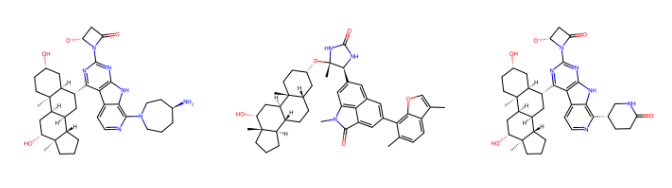
Toutes ces molécules possèdent le fragment avec 4 cycles non aromatiques.
Etape_2: Comparaison avec Ambinter
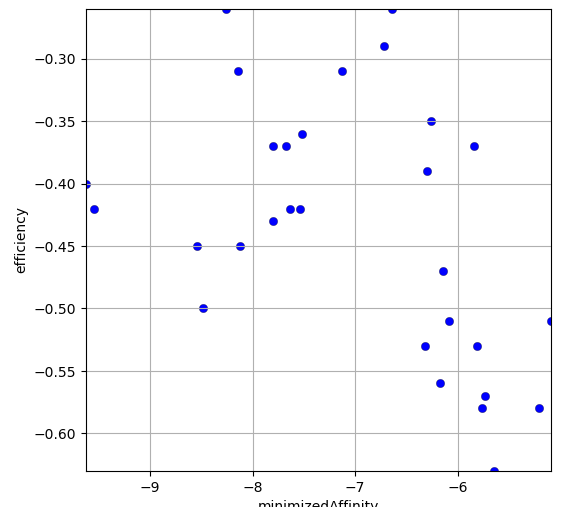
La comparaison de ces molécules générées avec la base Ambinter de Greenpharma a permis de trouver 13 728 molécules similaires qui après docking donnent le graphique suivant:
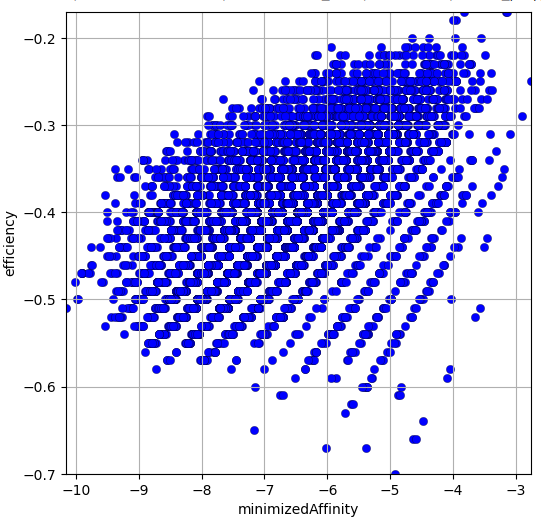
Quelques exemples de molécules avec un mimizedAffinity < -9.5:
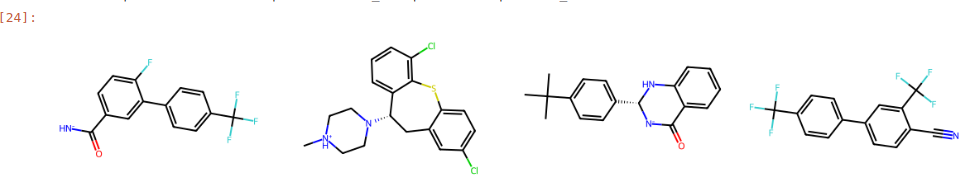
Quelques images dans le site:
 |  |
 |  |
Etape_3:Comparaison avec les molécules de la Chembl.
Il existe 29 molécules différentes dans la Chembl qui ont été testées sur la Beta-xylosidas, le docking de ces dernières donne le résultat suivant:
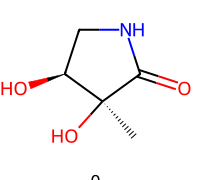 CHEMBL372907 | Seule molécule qui possèdent une efficiency proche de celle de la beta-D-xylopyranose. Son activité n’a pas pu être mesurée. |
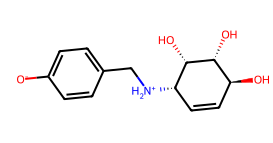 CHEMBL190256 CHEMBL190256‘minimizedAffinity’: ‘-8.12533’ ‘efficiency’: ‘-0.45’ ‘clogp’: ‘-2.16’ | Seule molécule ayant un pouvoir d’inhibition. |
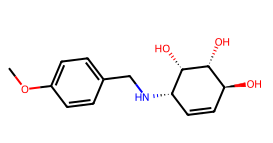 CHEMBL191983 ‘minimizedAffinity’: ‘-8.53777’ ‘efficiency’: ‘-0.45’ ‘clogp’: ‘-0.19’ | Molécules très similaire à la précédente mais qui n’a pas de pouvoir d’inhibition. |
Présentation d’Emolgine 0.5-1
La version Emolgine 0.5-1 permet de commencer à réfléchir aux prochaines étapes de son développement.
Emolgine 0.5-1 permet de répondre aux questions suivantes:
- Montre-moi des molécules ayant de l’affinité pour un site biologique d’une protéine dont le nom de gène commence par A, B, C
- Regroupe-moi les molécules par similarité.
- Montre-moi des molécules ayant de l’affinité pour au moins 5 sites différents.
- Montre-moi un réseau de sites avec des molécules ayant de l’affinité pour au moins 3 de ces sites
Les molécules peuvent être sélectionnées en fonction de différents paramètres.
Prochaines étapes:
Version 0.5 – 21/08/2024
Plusieurs avancées dans cette version.
Filtre sur les propriétés des molécules clogP, nHA, nHD, nRot, sascore, TPSA et alert pour moduler les affichages dans D3.JS.
Menu contextuel dans D3.js permettant de naviguer dans les données et de visualiser les propriétés des molécules sélectionnées.
Visualisations graphiques sous forme d’histogramme des grandeurs pour connaitre la répartition des molécules selon les grandeurs observées.
Visualisation en 2d avec JSXGraph pour travailler sur un lot de molécules.
Visualisation en 3d avec JSMOL et possibilité de cumuler des molécules pour comparaison.
La gestion des annotations personnelles ou communautaires associées aux molécules. Constitution d’une table dédiée pour ce stockage emolgine_annotations.
La gestion de la soumission des requêtes remontées au niveau de PHP et plus au niveau de JS pour des raisons de sécurité. Contrôle et conditionnement des requètes soumises sous NEO4J. Blocage des requêtes de type CREATE, DELETE, CALL et DROP (non autorisées)..
Le stockage systématique des requêtes exécutées dans une table MYSQL nommée emolgine_requetes doit permettre à terme de mettre en place un dashboard de suivi des performances, permettre également le partage entre utilisateurs et le développement d’un assistant.
Génération de peptides compatibles avec un site
Génération de peptides compatibles avec un site
emolgine permet de générer des ligands non peptidiques et des peptides.
Pour le moment, la décompostion des peptides de la PDB en acides aminés n’a pas été faite masi il faudra le faire.